Preview
논문에서는 whole slide image 암 진단 및 예후 예측에 많이 활용되고 있는 분야라고 설명을 하고 있습니다. 병리학자들이 Whole Slide Image(WSI)로부터, 정확하고 신뢰할 수 있는 진단 결과를 얻기 위해서는 많은 경험과 노력이 필요하며, WSI의 거대한 크기와 이질적인 특징들은 병리학적 판독 과정에 시간적인 소모가 많이 소요됩니다.
본에서는 Visual Question Answering(VQA) 방법을 통해 WSI를 해석하는 새로운 프레임워크인 WSI level의 VQA를 제안한다고 합니다.
WSI-VQA는 다양한 종류의 WSI level에 대한 질문을하고 응답을 받는 형태로 병리학자들이 인간 - 기계의 상호 작용을 통해서 등급을 분류한다던가 생존 예측을 한다던가 종양의 하위 유형을 분류를 수행 할 수 있는 범용성을 보여주고 있다고 합니다.
현재 데이터 셋은 997개의 WSI와 8672개의 WSI level의 질문 응답 쌍 포함하는 데이터셋을 구축했다고 합니다.
Wsi2Text Transformer(W2T)라는 이름의 생성적 모델은 기존의 판별적 모델보다 의학적 정확성에서 더 나은 성능을 발휘하며, 이는 임상 환경에서 모델이 적용될 가능성을 보여줍니다.
추가적으로, 진단 결과에 대한 text embedding과 WSI level에 visual embedding에 대한 co-attention mapping을 통해서 진단을 수행 했다고 합니다.
데이터셋과 코드는 해당 github https://github.com/cpystan/WSI-VQA 에서 확인할 수 있습니다.
1. Introduction
WSI-VQA: 병리학적 이미지 분석을 위한 혁신적 질문-응답 모델
최근 Multimodal Large Language Models (MLLM), 예를 들어 GPT-4V, LLaVa, Qwen-VL 등이 시각-언어 작업에서 탁월한 성능을 보여주고 있습니다. 특히, 의학 분야와 같이 전문 지식이 요구되는 환경에서도 이 모델들은 뛰어난 성과를 냈습니다. 하지만 이러한 모델들은 **전체 슬라이드 이미지(WSI)**와 같은 매우 고해상도의 데이터를 다루는 데는 한계가 있습니다.
WSI와 VQA의 한계
일반적인 자연 이미지에서의 VQA(Visual Question Answering)는 256x256 크기의 이미지를 다루며, 각 이미지에 대해 간단한 시각적 설명을 제공합니다. 그러나 병리학적 이미지의 경우, **Multiple Instance Learning (MIL)**을 사용하여 WSI를 여러 패치로 나누고, 이를 기반으로 학습을 진행해야 합니다. 이는 종양과 같은 미세한 시각적 특징을 구별하거나 생존 예측과 같은 복잡한 작업을 수행하는 데 필수적입니다.
WSI-VQA: 새로운 패러다임 제안
이 논문에서는 이러한 문제를 해결하기 위해 WSI-VQA라는 새로운 프레임워크를 제안합니다. 이 프레임워크는 WSI를 입력으로 받고, 질문-응답 형식으로 결과를 생성합니다. Visual encoder를 통해 이미지 패치 임베딩을 추출하고, text encoder를 통해 질문의 임베딩을 얻습니다. 이후 co-attention 메커니즘을 통해 패치와 텍스트 임베딩 간의 상호작용을 학습해 자유롭게 답변을 예측할 수 있습니다.
이 모델은 특히 후보 답변이 없는 임상 환경에서도 효과적입니다. 이전 VQA 방식이 단순히 정답을 선택하는 것과 달리, WSI-VQA는 단어 단위로 답변을 생성하여 더욱 복잡한 문제를 처리할 수 있습니다.
WSI-VQA의 한계와 데이터셋 문제
논문에서는 MLLM 훈련을 위한 데이터셋 부족과 WSI의 큰 해상도에서 발생하는 시각적 어려움, 그리고 데이터 프라이버시 문제를 언급하고 있습니다. 이를 해결하기 위해, WSI-VQA 데이터셋은 977개의 WSI와 8672개의 질문-응답 쌍을 포함한 데이터셋을 구축했습니다. 이 데이터는 TCGA(가장 큰 암 데이터베이스)에서 가져온 병리학적 텍스트를 기반으로 생성되었습니다.
WSI-VQA의 성능과 기여
실험 결과, WSI-VQA는 다양한 임상 작업을 통합하여 기존 MIL 방법보다 더 뛰어난 성능을 보였습니다. 특히, 조직 하위 유형 분류, 생존 예측, 면역조직화학적 등급 분류에서 우수한 성능을 입증했습니다.
논문의 기여
- WSI-VQA 프레임워크 제안: 병리학 분야의 다양한 슬라이드 수준 작업을 질문-응답 형식으로 재구성하여, 여러 작업에서 우수한 성능을 보여주었습니다.
- WSI-VQA 데이터셋 구축: 977개의 WSI와 질문-응답 쌍을 포함한 데이터셋을 공개하여, 병리학적 시각-언어 학습의 발전을 촉진할 것입니다.
- co-attention visualization: 모델의 해석 가능성을 높여, 병리학자들이 진단 과정을 더 잘 이해하고 활용할 수 있도록 돕습니다.
2. Dataset Construction
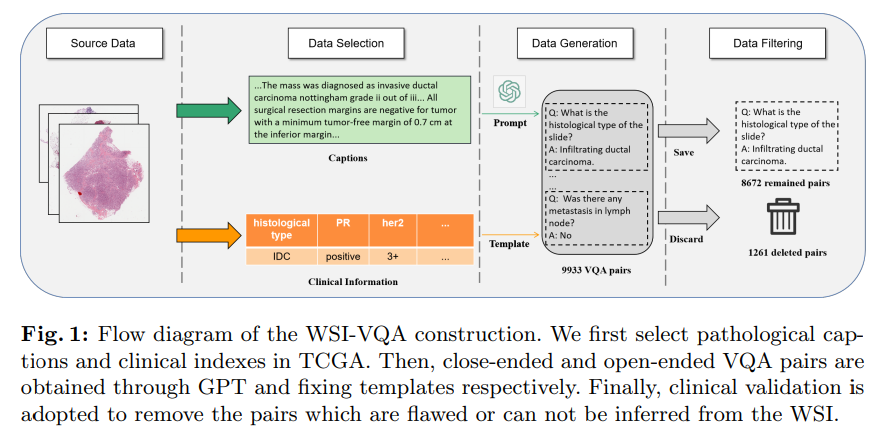
그림 1에서는 WSI-VQA 데이터셋의 구축 과정에 대한 자세한 내용에 대해서 나와있다고 합니다.
2.1 Data Generation
논문에서는 TCGA는 대규모 암 샘플을 수집하고 특성화하는 인프라를 구축했다고 하며, 이 인프라는 진단용 WSI와 다양한 방식의 관련 임상 정보를 포함하고 있다고 합니다. 본 논무에서는 신뢰성과 보안성 때문에 TCGA의 가장 큰 유방암 관련 하위 집합인 TCGA-BRCA를 사용하여 WSI- Level의 VQA 데이터셋을 구성했습니다.
그림 1에 표시된 것처럼, TCGA의 캡션에서 LLM(대형 언어 모델)의 도움을 받아 여러 선택지가 포함된 질문-응답 쌍을 생성했다고 합니다. 나머지 질문-응답 쌍은 TCGA의 임상 파일에서 키워드를 추출해 템플릿에 맞추는 방식으로 데이터를 생성 했다고 합니다.
Close-ended Subset
TCGA 의 병리학 보고서를 요약한 가장 큰 WSI 캡션 데이터셋을 기반으로, GPT와 같은 대형 언어 모델(LLM)의 도움을 받아 각 캡션에 대해 여러 질문과 후보 답변을 자동으로 생성했고, 다음과 같은 프롬프트를 사용하여 WSI-VQA 쌍을 생성했다고 합니다.
Ask 6 questions about the content and generate four options for each question.
The output should use the following template: i:‘the question index’ question:‘the
generate question’ choice: ‘A: option content B: option content C: option content
D: option content’ answer: The correct option.이 방법은 VQA 쌍 생성을 자동화하는 데 유용하지만, LLM에서 발생할 수 있는 환각(hallucination) 문제를 완전히 무시할 수 없다는 단점이 존재한다고 합니다. "환각이란 LLM이 상식에 어긋나거나 제공된 내용에 충실하지 않은 정보를 생성하는 현상을 말합니다."
따라서 논문에서는 데이터 필터링을 통해 심각한 결함이 있는 쌍을 제거했다고 합니다. 또한 WSI에서 유추할 수 없는 답변도 수집된 데이터셋에서 제외 해다고 합니다. 그 결과, 질문과 답변뿐만 아니라 후보 선택지도 포함된 총 4535개의 Close-ended Subset VQA 쌍을 얻었다고 합니다.
Close-ended Subset은 각 질문에 대한 선택지가 있으므로, 생성 모델의 성능을 평가하기 위해 의료적 정확도를 계산할 수 있다고 합니다.
Open-ended Subset
TCGA-BRCA에는 하위 유형 분류, 면역조직화학적 테스트, 생존 예측과 같은 다양한 임상 지표가 포함되어져 있어서 질문-응답 템플릿에서 키워드와 해당 값을 일치시켜, 풍부한 임상 정보를 포함하는 고품질 VQA 쌍을 얻을 수 있습니다. 논문에서는 다음과 같은 templete을 적용해서 질문 - 응답 데이터 쌍을 얻었다고 합니다.
Q: What is the result of [KEY]? A: [VALUE].훈련 과정에서는 각 쌍에 대해서 무작위로 템플릿을 선택하돌록 했고, 생성 모델이 합리적으로 작동하도록, 진단용 WSI에서 유추할 수 없는 임상 지표는 제외하였습니다. 그 결과, 개방형 하위 집합에는 총 4137개의 VQA 쌍이 포함 되어져 있다고 합니다.
Data Statistics.
생성한 데이터 셋은 총 997개의 WSI와 8672개의 질문-응답(QA) 쌍으로 구성되어져 있다고 합니다. 각 WSI는 평균적으로 8.9개의 QA 쌍과 연결이 되어져 있으며, QA 쌍 중에서, Close-ended Subset 에는 4535개의 QA 쌍이 있고, Open-ended Subset 4137개의 QA 쌍이 포함되어 있습니다.

Fig. 2 (a)에 나타난 바와 같이, 데이터셋의 질문은 경계 상태 식별과 같은 공간 정보를 요구하는 질문부터 WSI의 면역조직화학적 등급 분류와 같은 도전적인 질문까지 다양성을 보여줍니다.

Fig. 2 (b)는 질문의 다양한 카테고리 분포를 보여줍니다. 데이터셋의 주요 질문 유형은 'what' 질문으로, 전체 내용의 80%를 차지합니다. 그 외에도 13.2%의 '예/아니오' 질문, 1.4%의 'where' 질문, 3%의 'which' 질문이 있습니다.

Fig. 2 (c)에서 각 엔터티의 커버리지 비율을 계산했습니다. 예를 들어, 'her2'의 커버리지 비율은 약 50%로, 이는 Open-ended Subset의 절반 정도의 사례에서 Her-2 결과가 제공되지 않았음을 의미합니다. 즉, HER2에 대한 open-ended subset이 완전히 없는 것은 아니지만, 절반의 사례에서만 HER2 결과가 제공하고 있다고 합니다.
3.Method

논문에서는 WSI-VQA 모델과 Wsi2Text Transformer(W2T)에 대해 설명하는 섹션이라고 합니다. Wsi2Text Transformer에 대한 내용은 그림3에 나와 있다고 합니다.
3.1 Problem Formulation
입력은 WSI인 X_i와 질문인 Q_i, 출력으로 예측할 답변 Y_i입니다. WSI의 경우 큰 해상도를 가지고 있으므로 전처리 관정이 필요하다고 합니다. 논문에서는 WSI를 작은 해상도를 가지는 패치 그룹으로 구성된 집합으로 처리했으며, 이는 다음과 같이 표현한다고 합니다.
WSI를 작은 해상도를 가지는 패치 그룹으로 구성된 집합:

X_i는 i번째 WSI이고, M_i는 시퀀스 길이로, 보통 10,000 이상입니다. 패치 임베딩을 추출하기 위해 시각 추출기 h를 사용하며, 이는 다음과 같이 표현할 수 있다고 합니다. :
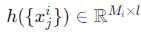
여기서 l은 임베딩 크기입니다.
질문의 경우, 토큰화 후 토큰 시퀀스로 볼 수 있으며, 다음과 같이 나타 낼 수 있다고 합니다.
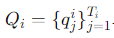
여기서 T_i는 토큰의 개수입니다. 우리는 텍스트 추출기 g를 사용하여 토큰을 단어 임베딩으로 변환하며, 이는 다음과 같이 나타 낼 수 있다고 합니다.:

여기서 k는 단어 임베딩의 크기입니다.
생성 모델은 답변은 다음과 같이 표현이 된다고 합니다.
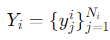
여기서 N_i는 타겟 시퀀스의 길이입니다. 이 모델은 시퀀스에서 단어들의 조건부 확률 합을 극대화하는 언어 모델 손실 목표로 훈련됩니다.
손실 함수는 다음과 같이 나타낼 수 있습니다:

3.2 Preprocessing for Bag Construction
Visual Embeddings.
자연 이미지에서 VQA(Visual Question Answering) 작업은 GPU에서 이미지를 직접 입력하여 시각 임베딩을 학습할 수 있습니다. 그러나 병리학에서는, 큰 이미지를 작은 개별 패치로 분할하여 처리하는 MIL 방식을 따릅니다. 먼저 전처리 작업을 수행하여, 전체 슬라이드 이미지에서 정보가 없는 영역(일반적으로 전체 이미지의 70%를 차지하는 부분)을 제거합니다.
구체적으로, WSI에서 낮은 해상도(작은 level)에서 RGB 색 공간에서 HSV 색 공간으로 변환됩니다. 그 후, 채도 채널을 기반으로 흰색 영역을 필터링합니다. 그런 다음, 전경 영역에서 슬라이딩 윈도우 방식을 사용하여 겹치지 않게 패치를 자릅니다.
패치를 얻은 후 사전 훈련된 신경망을 사용하여 이러한 패치에서 특징을 추출합니다. 256×256 크기의 패치는 ResNet을 사용하여 512차원 시각 임베딩으로 변환되며, 이는 계산 복잡성을 크게 줄입니다. 주목할 점은, 시각 추출기는 학습되지 않는다는 것입니다.
Word Embeddings
질문 시퀀스는 시각 임베딩보다 훨씬 짧습니다. 질문의 평균 시퀀스 길이는 9.2개의 토큰이며, 일부 질문은 최대 24개의 토큰을 포함할 수 있습니다.
따라서 단어 임베딩은 처음부터 학습하는 것이 가능합니다. 우리의 실험에서는, BioClinicalBert와 PubMedBert라는 두 가지 잘 정립된 언어 모델을 텍스트 추출기로 사용했습니다. 이 모델들은 풍부한 의료 데이터를 기반으로 훈련되어 우수한 단어 임베딩을 제공합니다.
3.3 Interaction between WSI and Text
현재 병리학 분야에서의 다중 모드 접근법은 서로 독립적인 패치와 해당 텍스트 설명을 연결하는 데 집중하고 있습니다. 하지만 이러한 방식은 WSI가 여러 관련 패치들의 집합임을 고려하지 않고 패치 수준의 설명에 집중하기 때문에, WSI - level의 작업에서 한계가 있습니다. 우리 프레임워크에서는 WSI와 텍스트를 bag representations로 모델링하여, 동일한 WSI에서 잘라낸 패치들 간의 상호작용을 모델링할 뿐만 아니라, 패치 토큰과 단어 토큰 간의 세밀한 상호작용도 설정할 수 있습니다.
시각 임베딩 EX ∈ RMi×l을 얻은 후, 우리는 transformer encoder를 사용하여 인스턴스 간의 상호 관계를 캡처합니다. 이는 다음과 같이 수식화됩니다:

여기서 W_Q,W_K,W_V∈Rl×l은 학습 가능한 매개변수이고, d는 스케일링 인자입니다. Transformer 인코더 T_e에서 모델은 인스턴스 간의 상호 관계를 학습하여 이후의 디코딩 단계에서 사용할 상호 인스턴스 상관성을 구축합니다.
이 과정은 병리학자가 보통 특정 관심 영역을 관찰하고 세포 수준의 특징과 조직 및 기관의 구조적 정보를 바탕으로 진단을 내리는 임상 워크플로우를 모방한 것입니다.
또한, transformer decoder에서는 co-attention 메커니즘을 채택하여 시각적 개념과 텍스트 개념 간의 관계를 캡처합니다. 이는 다음과 같이 수식화됩니다:

여기서 W_Q,W_K,W_V는 학습 가능한 행렬이고, 단어 임베딩을 k에서 l로 변환하기 위해 선형 매핑 T_l을 거칩니다. co-attention 모듈 A∈RT_i×M_i는 단어 토큰 q_i가 패치 x_i에 얼마나 주의를 기울이는지 보여주는 패치-단어 유사성을 학습합니다.
각 WSI-질문 쌍에 대해, 우리는 질문에서 생물학적 엔터티를 키워드로 선택하여 답변에 대한 시각적 설명으로 주의 맵을 생성합니다. 예를 들어, 질문에서 'her2'가 4번째 토큰인 경우, 우리는 주의 맵으로 A4∈R1×Mi를 선택해 시각화합니다.
논문에서는 co-attention 맵이 모델이 특정 단어와 관련된 패치에 얼마나 주의를 기울였는지를 시각적으로 보여줄 수 있다고 합니다.
4. Experiments
해당 섹션에서는 논문에서 제안한 모델인 W2T의 설정에 대해 설명을 한다고 합니다.
4.1 WSI-VQA 데이터셋
우리가 제안한 WSI-VQA 데이터셋에는 총 977개의 WSI와 8671개의 질문-응답 쌍이 포함되어 있습니다. 데이터셋을 훈련, 검증, 테스트 세트로 분할했습니다. 훈련 세트는 804개의 WSI와 7139개의 질문-응답 쌍을 포함하고, 검증 세트는 87개의 WSI와 798개의 쌍을 포함하며, 테스트 세트는 86개의 WSI와 735개의 질문-응답 쌍을 포함한다고 합니다.
Close-ended Subset 테스트 세트에서는 각각 A에 151개, B에 107개, C에 86개, D에 46개의 정답 선택지가 있습니다. 우리는 모델 성능을 평가하기 위해 언어 지표와 임상 지표를 모두 사용했습니다. Close-ended Subset 에서는 모델 성능을 측정하기 위해 문장 유사성을 비교하여 정확도(ACC)를 사용했다고합니다.
4.2 Visual Extractors
이 섹션에서는 생성 모델에 사용된 다양한 시각적 추출기를 소개합니다. ResNet [15]은 ImageNet에서 많은 자연 이미지와 분류 레이블로 사전 훈련된 고전적인 비전 모델을 사용했고, 또 본 논문에서는 사용하는 버전은 ResNet-50으로, 50개의 컨볼루션 층과 25.6M 파라미터로 구성되어 있다고합니다.
ViT [11]는 Vision Transformer로, VIT-S 버전을 사용했으며 약 22M 파라미터를 가진 12개의 Transformer 층으로 이루어져 있습니다.
DINO [5]는 표준 ViT 모델을 사용하여 자가 지도 학습 방식으로 사전 훈련된 전략입니다. 우리는 VIT-S를 백본으로 선택해 TCGA-BRCA에서 자른 패치를 사용해 사전 훈련했습니다.
HIPT [7]는 피라미드 트랜스포머를 사용해 계층적 자가 지도 학습을 수행하며, WSI에 특화된 사전 훈련 방식입니다. HIPT는 WSI에 내재된 계층적 구조를 활용해 고해상도 이미지 표현을 학습했다고합니다.
4.3 Text Extractors
text 임베딩 모델은 크게 BioClinicalBert과 PubMedBert, Scratch를 사용했다고 합니다.
BioClinicalBert : BioClinicalBert [1]는 BERT와 유사한 모델로, MIMIC III [18] 데이터베이스의 모든 기록에서 훈련
PubMedBert : PubMed 및 PubMedCentral [34]에서 훈련된 모델로 BERT-Base 구조에 기반하여 3.1B 단어로 훈련
Scratch : 임베딩 매핑을 사용해 텍스트 특징을 추출하되, 파라미터를 고정하지 않는 방법을 의미합니다
단어 토큰의 길이가 이미지 토큰보다 훨씬 짧기 때문에, 텍스트 추출기를 처음부터 학습하는 것이 가능하다고 합니다.
4.4 Training Setting
논문에서는 transformer encoder와 decoder를 3개의 층으로 구성했다고 하며, hidden size의 경우, 512라고 합니다. multi-head self-attention 4개의 head를 가지고 있다고 합니다.
모델을 최적화 시키기 위한 옵티마이저는 adam을 사용해 학습을 진행 했고, lr은 1e-4로 설정했다고 하며, weight decay 5e-5라고 합니다.
inference시에는 beam search의 크기를 3으로 설정 했다고 합니다. pytorch를 사용해서 진행 했고, GPU는 A100 - 80GB 모델을 사용했다고 합니다.
5.Results
5.1 Benchmark

논문에서는 벤치마크 성능을 측정하기 위해 두 가지 종류의 지표를 고려했다고 합니다. :
- 자연어 생성(NLG) 지표로, BLEU, METEOR, ROUGE 등이 포함되며, 이는 자연어 처리 작업에서 널리 사용됩니다.
- 모델의 임상 성능을 측정하기 위한 실제적 일관성 및 완전성(FCC) 지표
Close-ended Subset에서는 다수의 후보 중 하나를 정답으로 선택해야 하므로 정확도(ACC)를 사용해 성능을 평가했으며, Factent는 병리학적 도메인에서 생성된 엔터티가 참조와 얼마나 일치하는지를 측정합니다.
Visual extractor를 Resnet 일 경우에 NLG 지표에서 가장 좋은 결과는 텍스트 추출기를 처음부터 학습했을 때 달성되었습니다. 특히, PubMedBert를 사용한 모델과 비교했을 때, WSI-VQA에서 BLEU-1에서 +2.4, BLEU-4에서 +0.6, METEOR에서 +1.2, ROUGE에서 +3.0의 성능 향상이 있었다고 합니다.
처음부터 학습한 텍스트 추출기는 더 유창하고 읽기 쉬운 답변을 생성하는 데 있어 우수한 성능을 보였지만, 사전 훈련된 임베딩을 사용하는 경우 NLG 지표에서 살짝 아쉬운 성능을 보였다고 하며, 이는 사전 훈련에 사용된 코퍼스와 우리의 병리학적 코퍼스 간의 도메인적 차이 때문일 수도 있다고 합니다.
그러나 'Scratch' 전략은 FCC 지표에서 일관되게 우세하지는 않았습니다. 예를 들어, BioClinicalBert가 ViT에서 제공된 시각적 임베딩과 함께 사용될 때 ACC에서 +4.1, Factent에서 +2.1의 증가를 보였으며, 이는 잘 정립된 텍스트 추출기가 임상 개념을 인식하고 이해하는 데 잠재력을 가짐을 보여준다고 합니다.
Visual extractor에 관해서는, DINO와 HIPT(도메인 내 사전 훈련)가 의료적 정확성에서 일관되게 ResNet과 ViT(도메인 외 사전 훈련)보다 좋은 성능을 보이는 것을 관찰할 수 있었습니다.
특히, DINO 사전 훈련 추출기를 채택했을 때 ViT를 사용할 때와 비교해 ACC에서 +2.4, +1.4, +5.4의 큰 증가를를 보였으며, 도메인 내 데이터로 사전 훈련된 시각적 추출기가 병리학적 패치에서 관련 특징을 더 잘 추출할 수 있으며, 이는 WSI의 진단에 크게 기여한다는 것이 합리적입니다. 모든 벤치마크에서 ACC 지표는 거의 50에 가깝고, Factent 지표는 90 이상으로, WSI-VQA가 임상 환경에서 잠재력을 가지고 있음을 보여준다고 합니다.
5.2 Slide-level Tasks
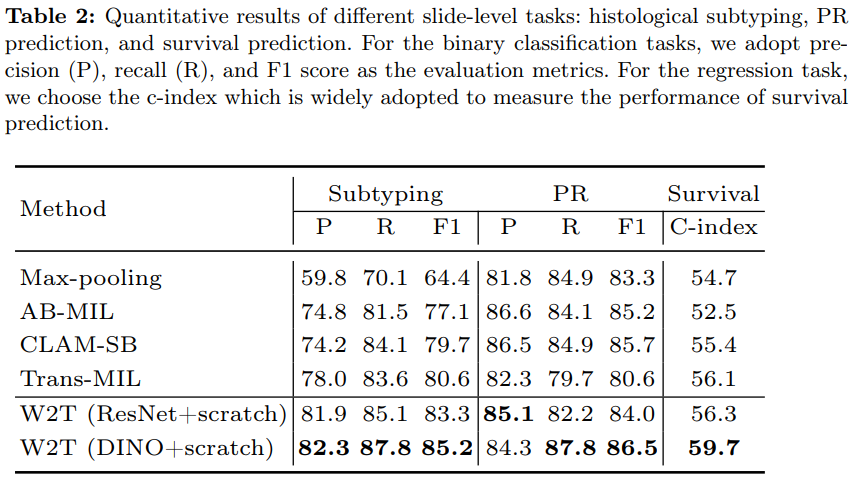
최근 몇 년 동안 AI 기반의 디지털 병리학이 발전하면서, 일부 딥러닝 모델은 슬라이드 수준 작업에서 인간 수준의 성능을 달성하기도 했습니다.
우리의 WSI-VQA 방법은 다양한 슬라이드 수준 작업을 다룰 수 있으며, 적절한 프롬프트를 제공하면 여러 종류의 임상 작업을 처리할 수 있습니다. 따라서 우리는 제안한 모델의 성능을 특정 임상 작업에서 평가하고, WSI 분류에서 널리 사용되는 다중 인스턴스 학습(MIL) 방법과 비교했다고 하며, 그 결과는 표 2에 나와 있습니다. MIL 접근법은 WSI를 처리하기 위해 정교하게 설계된 방법으로, 여기에는 AB-MIL [9], CLAM-SB [27], TransMIL 등을 W2T 프레임워크는 모든 MIL 접근방법을 크게 능가 했다고 합니다.
ResNet을 사용하는 모델은 두 번째로 좋은 MIL 방법과 비교하여 정밀도(precision)에서 +3.9, 재현율(recall)에서 +1.0, F1 점수에서 +2.7만큼의 성능 향상을 보였다고 하며, 만약 DINO로 사전 훈련된 시각적 추출기를 사용하면 F1 점수가 85.2로 더 향상된 성능을 보여준다고 합니다.
PR 예측은 내분비 치료의 기초가 되는 호르몬 수용체 예측을 하는 Task에서도 PR 예측 작업에서도 우리의 모델은 MIL 방법들과 비교하여 경쟁력 있는 성능을 보였다고 합니다.
생존 예측은 WSI에서 복잡한 특징을 포착해야 하며, 순서 회귀 작업이기 때문에 계산 병리학에서 매우 까다로운 작업입니다. ResNet 또는 DINO를 사용한 우리 모델은 각각 C-Index에서 56.3 및 59.7의 성과를 달성했습니다.
요약하자면, WSI-VQA는 조직 하위 유형 분류, PR 예측, 생존 예측과 같은 다양한 슬라이드 수준의 임상 작업에서 뛰어난 성능을 발휘했으며, 특히 DINO 시각 추출기와 결합된 경우 더 높은 성과를 기록했다고 합니다.
5.3 Visualization

시각적 개념과 단어 임베딩 간의 상호작용을 반영하는 co-attention 히트맵은 의사결정 과정을 직관적으로 설명할 수 있으며, 병리학자들에게 임상적인 단서를 제공할 수 있다고 합니다.
논문에서는 질문에 포함된 키워드에 주의를 기울이는 co-attention 가중치를 해당 WSI의 썸네일과 오버레이(overlay)하여 시각화했다고 합니다. 높은 주의 가중치를 가진 이미지 패치들은 히트맵에서 빨간색으로 강조된다고 합니다.
그림 5에서 볼 수 있듯이, 우리는 두 가지 사례에서 네 개의 co-attention map을 선택해 사용했다고 하며, 생성 모델이 질문에 따라 관심 있는 영역에 주의를 기울일 수 있다는 것을 관찰할 수 있었다고 합니다.
예를 들어, "margin status"에 대한 질문이 주어졌을 때, 모델은 종양의 경계에 집중했다고 하며, "종양 크기(mass size)"에 대한 질문이 주어지면, 모델은 슬라이드 전체의 종양에 주의를 기울였다고 합니다.
이러한 시각화 결과는 모델의 공간적 인식을 반영한다고 하며, 면역조직화학(IHC) 작업에서는, 모델이 고등급 종양 형태를 나타내는 영역보다 종양 관련 기질에 더 많은 주의를 할당하는 것을 볼 수 있었다고 합니다.
PR 예측에서는, 높은 주의가 할당된 영역이 종양 세포와 미세 환경(예: 종양 기질)에 집중되었지만, Her-2 결과를 예측할 때는 무시되었다고 합니다.
임상 환경에서는 H&E 이미지를 기반으로 직접적으로 Her-2와 같은 IHC 결과를 얻을 수 없지만, 최근의 연구들은 오직 WSI만을 가지고도 Her-2 상태를 예측할 수 있다는 것을 입증했습니다.
이러한 연결은 추가적인 연구가 필요하지만, 우리의 방법은 관련된 임상적 단서를 제공할 수 있으며, AI 접근 방식을 병리학자들이 더 잘 이해할 수 있도록 해줄 줄 것을 기대한다고 합니다.
6 Limitations and Future Work
비록 WSI-VQA가 다양한 슬라이드 수준 작업에서 높은 성능을 보였지만, 여전히 몇 가지 한계와 향후 연구의 여지가 있습니다.
- 데이터셋의 제한성: 현재 WSI-VQA 데이터셋은 주로 TCGA-BRCA에 기반하고 있으며, 다른 암 유형이나 조직(예: 폐암, 대장암)과 같은 다양한 임상 시나리오를 포함하지 못하고 있습니다. 향후 연구에서는 다양한 병리학적 상황을 다룰 수 있는 더 포괄적인 데이터셋 구축이 필요합니다.
- 모델 해석력의 한계: **공동 주의 맵(co-attention map)**을 통해 모델이 진단 의사결정을 시각적으로 설명할 수 있지만, 여전히 임상적으로 정확한 해석을 제공하는 데 한계가 있습니다. 모델이 특정 패치에 주의를 기울이는 이유를 더 잘 설명할 수 있는 개선된 방법이 필요합니다.
- 임상 환경에서의 성능: 생성 모델을 사용하여 임상 정보를 예측하는 것은 매우 정확한 결과를 요구하는 실제 임상 환경에서 충분하지 않을 수 있습니다. 따라서, 더 높은 정확성을 목표로 하고, 다양한 임상 작업을 처리할 수 있는 모델을 개발하는 것이 중요합니다.
결론적으로, WSI-VQA는 병리학적 이미지 분석에서 새로운 가능성을 열었으며, 향후 연구를 통해 더 포괄적이고 정확한 모델로 발전할 수 있을 것입니다.
7 Conclusion
본 논문에서는 WSI-VQA라는 새로운 프레임워크를 제안하여 전체 슬라이드 이미지(WSI)에서 다양한 병리학적 작업을 수행할 수 있는 질문-응답(QA) 방식을 도입했습니다.
이를 통해 면역조직화학적 등급 분류, 생존 예측, 종양 하위 유형 분류와 같은 중요한 작업들을 효과적으로 처리할 수 있음을 입증했습니다. 또한, 977개의 WSI와 8672개의 질문-응답 쌍을 포함한 WSI-VQA 데이터셋을 구축하여, 모델의 성능을 평가할 수 있는 벤치마크를 제공했습니다.
실험 결과, 우리의 Wsi2Text Transformer(W2T) 모델은 기존 다중 인스턴스 학습(MIL) 모델들보다 더 높은 정확성과 임상적 일관성을 보여주었으며, 이는 병리학적 이미지 분석에서 매우 유용할 가능성을 시사합니다. 또한, co-attention 맵을 시각화하여, 모델이 진단 결과를 도출하는 과정을 직관적으로 설명할 수 있음을 확인했습니다. 이는 병리학자들에게 중요한 임상적 단서를 제공하고, AI 모델의 해석 가능성을 높이는 데 기여합니다.
앞으로, 더 다양한 암 유형과 조직을 포함한 데이터셋 확장, 더 높은 진단 정확성을 목표로 한 모델 개선, 그리고 AI 모델의 투명성과 해석 가능성을 높이는 연구를 계속 진행할 계획입니다. 이를 통해 WSI-VQA는 병리학적 이미지 분석에서 중요한 도구로 자리 잡을 것입니다.
'논문 정리' 카테고리의 다른 글
| [논문 리뷰] Denoising Diffusion Probabilistic Models (7) | 2025.06.01 |
|---|---|
| [간단리뷰]Unet++ (0) | 2024.05.11 |
| [review]PETALS: Collaborative Inference and Fine-tuning of Large Models (0) | 2023.08.08 |
| YOLOV1 (0) | 2023.07.02 |
| Faster R-CNN (0) | 2023.06.24 |



